前回、Alphefold2でアミノ酸配列からタンパク質の立体構造を予測したが、今回は逆問題として立体構造からアミノ酸配列を予測するということで、タンパク質MPNNを試したみた。
タンパク質MPNN
タンパク質の骨格が決まったときに、その構造に折り畳まれるアミノ酸配列を見つけることも課題となる。Rosettaのようなこれまでの手法はエネルギーを計算して最も低いエネルギーを探索していたが、今回試してみたタンパク質MPNN(以下、ProteinMPNN)ではディープラーニングのアプローチで原子座標からアミノ酸配列を予測する。側鎖の回転状況を明示的に考慮しなくても、アミノ酸配列を高速に予測できると期待されている。(Protein MPNNの元論文はこちら)
その元となっているのは、MPNN(message passing neural network)というグラフニューラルネットワーク(GNN)。エッジとノードで表現されたグラフデータを深層学習で扱う手法。グラフデータは普段は馴染みがないが、SNSの友人関係、路線図、化合物の化学構造などを表現するのに使われる。
画像データと同じようにグラフ構造データも畳み込みの概念が使われていて、グラフ畳み込みニューラルネットワーク(GCN)と呼ばれている。GCNの方法としては、ノードに情報を集約させて、更新して、読み出すというフレームワークで行われている(ここのサイトの非常に説明がわかりやすい)。この集約する部分がメッセージパッシングの考え方で、化合物だと隣接する原子間の接続情報などの情報を集約させている。
実験結果
以下のサイトからデモを行うことができる。サイトにある図のように簡単にPDBのデータを取り込んでProteinMPNNでアミノ酸配列を予測でき、しかも予測したアミノ酸配列を元にAlphaFold2で立体構造を復元して元の立体構造と比較することができる。
https://huggingface.co/spaces/simonduerr/ProteinMPNN
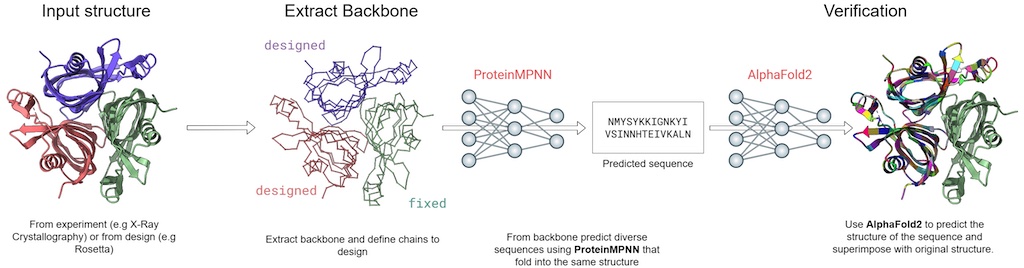
このデモ環境の使い方は非常に簡単で、InputのところにPDBの番号を入れて、Runボタンを押すと実行してくれる。下側にOutput欄があり、予測したアミノ酸配列が表示される。今回、ミオグロビン(PDB: 1MBN)を使ってみる。ミオグロビンは筋肉中で酸素を蓄える役割を持つタンパク質である。
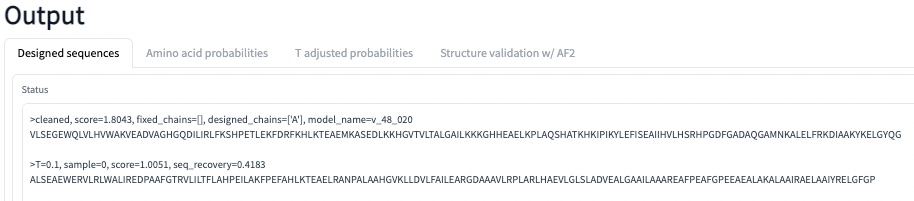
Inputに”1MBN”を入れて実行すると、数秒程度でOutputの”Designed sequences”でアミノ酸配列が表示される。2つアミノ酸配列が並んでいるうち、上側が正しい配列、下側が予測した配列である。seq_recoveryが0.4以上なので、まずまずの結果になった。
次に”Structure validation w/ AF2″をクリックするとAlphaFold2で立体構造予測をしてくれる。その結果が以下の図である。RMSD(平均二乗偏差)が1.28で良い結果ではないかと思う。(立体構造でグレーの部分は正解の立体構造、色があるのがAlphaFold2の結果)
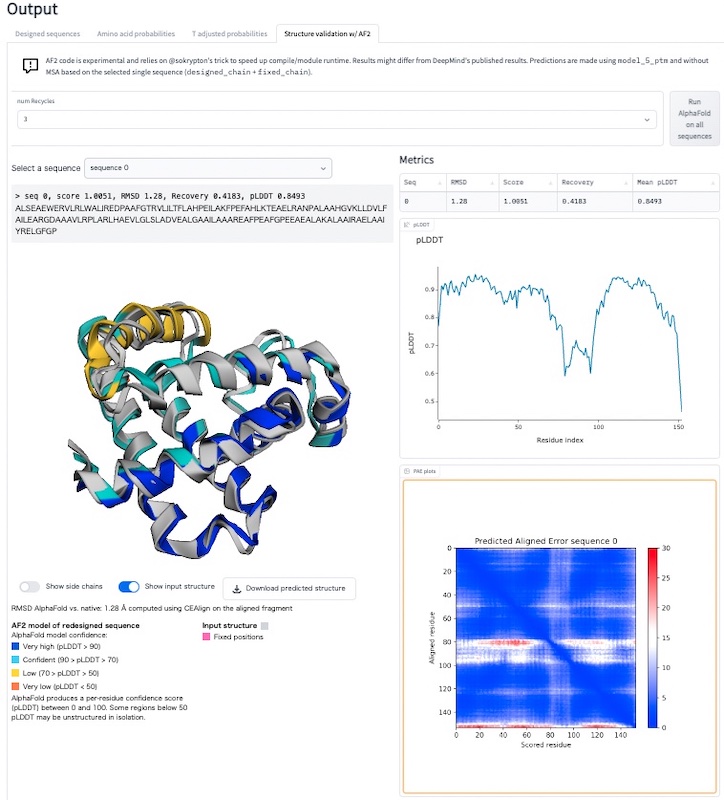
ミオグロビンでは良い結果が出たが、ここに至るまで色々なタンパク質をトライしたが、中々sequence_recoveryの値が高いものが得られなかった。デモ版でなく、コードから実行すれば違った結果が出るのかもしれないけど、ボタン2つ押すだけで、アミノ酸配列と構造予測をしてくれるので、便利な環境であることは間違いない。